基础概念
基因渐渗(introgression):,Introgression is the movement of a gene from one species into the gene pool of another by the repeated backcrossing of an interspecific hybrid with one of its parent species。关键点:属于两个物种之间的基因流动,通常是后代种与祖先种的反复回交实现祖先种的一些基因区段(表现为潜在的祖先性状)再次出现在后代种当中。
不完全谱系分选(ILS,Incomplete Lineage sorting):位点树(基因树)和物种树不一致的现象。例如ABC物种分化前某一位点存在多态性,为0/1。随着C分化出去,而此多态性的位点在C中发生了固定,为1,而在AB祖先中是以多态存在。当AB发生分化时,此等位以随机的方式分别进入AB物种中。当B的位点和C相同时,即发生BC关系更近的现象。我们以为是BC之间存在基因流的现象,而实际上为不完全谱系分选。
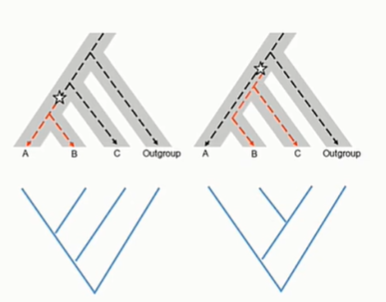
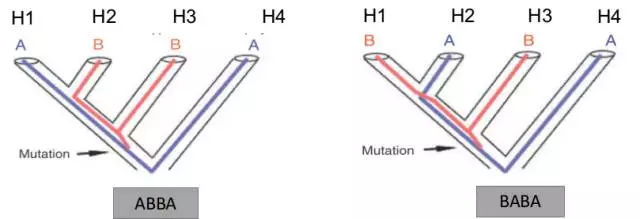
ABBA-BABA统计(D统计量):是研究基因渐渗的经典方法。在一个给定的拓扑关系(((H1,H2),H3),H4)。H1,H2是姊妹类群(多为一个物种内的两个种群),H3是潜在的基因渐渗来源物种,H4是外类群。假设给定祖先H4的位点为A,潜在渐渗祖先种H3位点为B。同时在H1,H2之间也存在allele的不同(AorB),则如果不存在H3的渐渗,则H1和H2的两个种群为AB或者BA的概率各为50%,而如果H3对H2存在有渐渗作用,则H2为B,H1为A的概率大于H2为A的概率。
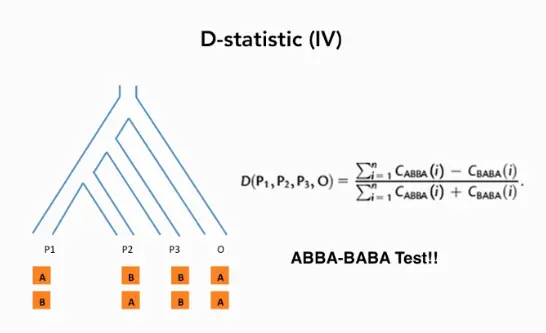
以下即为计算D-统计量的方法,当D值为0时,表示H1,H2与H3不存在渐渗,当D大于0,则表示ABBA的情况更多,表示H2和H3之间的关系相对于H1更近,即存在于H3到H2的渐渗。反之D<0 则表示H1和H3之间关系更近,存在H3向H1的渐渗。D值范围-1~1。
其它统计量还包括:partition D-statistic, 以及近期较为流行的fd统计值,fdm统计值等。
软件:Admixtools/admixr
软件Admixtools[https://github.com/DReichLab/AdmixTools]为计算渐渗杂交相关统计量软件,其软件统计算法基于Patterson et al. (2012) Ancient Admixture in Human History。软件主要包括6个程序:
convertf: VCF格式转换qpDstat: D检验,test of admixture with 4 popqp3Pop: f3检验qpBound: estimating bounds on the admixture proportions.qpF4Ratio: f4检验。rolloff: dating admixture events.
软件admixr[https://bodkan.net/admixr/articles/tutorial.html]为更好做admixtools分析的R包软件。
软件接受EIGENSTRAT格式的数据文件,分别为ind, snp, geno文件。我们通常使用的vcf需要进行转换。
实操
1. 数据准备
1 | library(admixr) |
下载生成snps.geno, snps.ind, snps.snp。其中ind文件为3列分别为:ind, sex, population。而第三列population为admixr使用的一列。
2. D检验
ADMIXTOOLS实际计算的是BABA-ABBA的值,所以D>0,表示H1和H3更为近,区别于ABBA-BABA的结果,与其相反。
1 | pops <- c("French", "Sardinian", "Han", "Papuan", "Khomani_San", "Mbuti", "Dinka") |
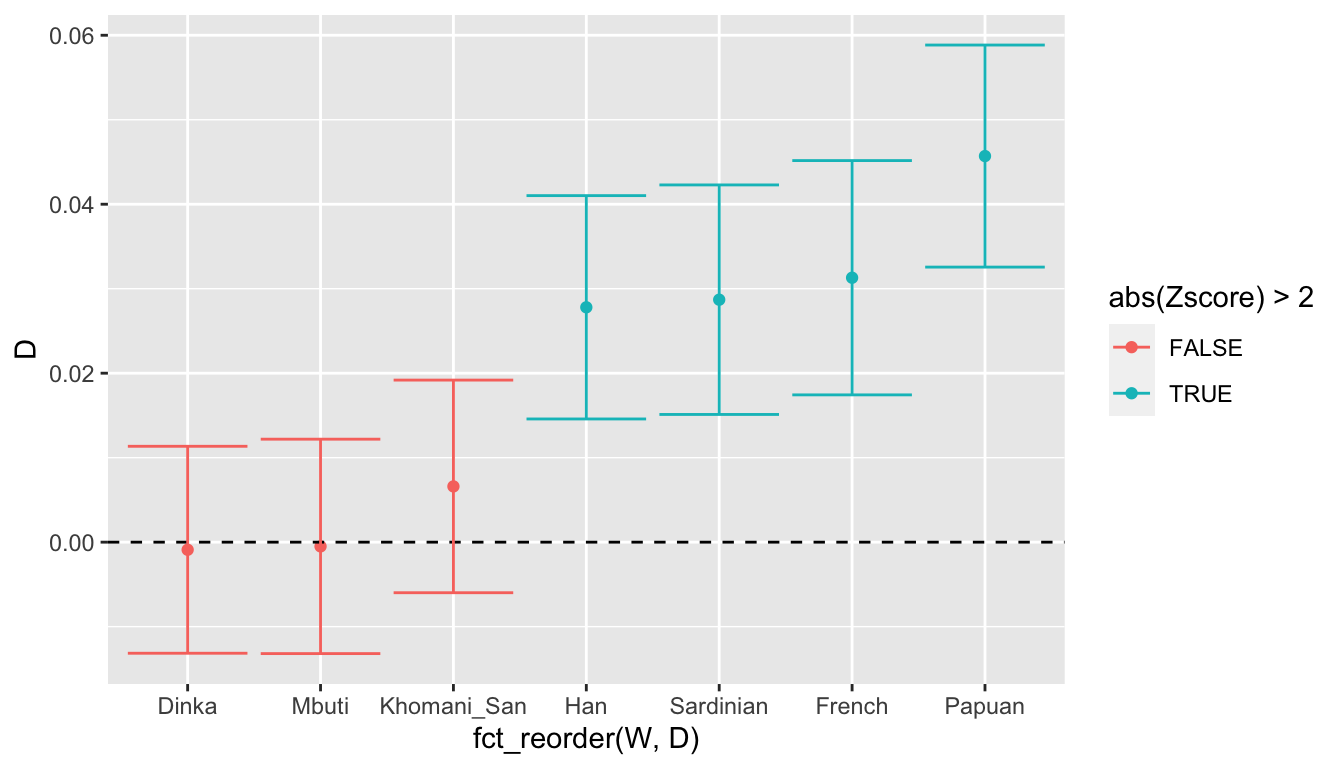
结果表示Y的尼安德特人对于3个非洲群体是D值趋于0,符合null hypothesis:无基因渐渗影响;而对于其它走出非洲的族群都存在有0.03左右的D值影响。说明尼安德特人对于走出非洲的族群存在有渐渗作用。
3. f4检验
This is a test of whether four taxa are two pairs of sister taxa ((W,X),(Y,Z))。
f4计算公式类似于D检验:当f4值>0表示存在W和Y或者X和Z之间的基因流。
1 | result <- f4(W = pops, X = "Yoruba", Y = "Vindija", Z = "Chimp", data = snps) |
D检验会受到外类群H4 outgroup的真实情况影响,当H4实际和H1/H2共享较多位点时,此时任意的H3都会算出D值>0的假阳性现象。因此,实际计算的过程中需要有副对照、正对照。设置一个不会发生渐渗的祖先群H3做对照。
4. f4-ratio检验
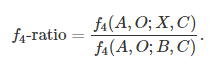
给定D值>0,H2已经受到了H3的渐渗作用影响,f4-ratio检验为探究H2受到了多少H3的血源成分。f4-ratio检验接受5个taxa:X, A, B, C, O。The introgressed taxon “X”, its sister taxon “C”, the source of introgression “B” and its sister taxon “A”, and an outgroup “O”.
1 | result <- f4ratio(X = pops, A = "Altai", B = "Vindija", C = "Yoruba", O = "Chimp", data = snps) |
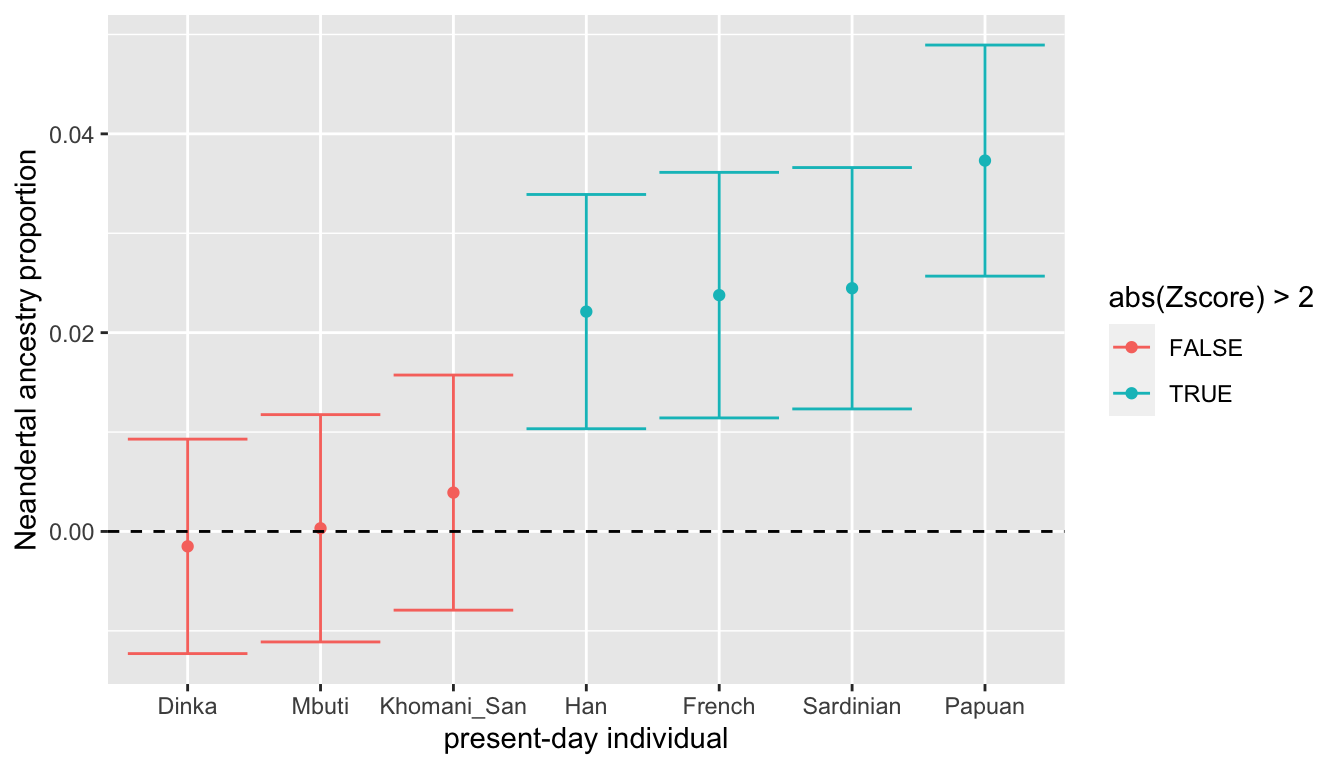
现代走出非洲的族群普遍有2~3%的尼安德特人学院成分。并且在Papuan族群中显示了更高的Neanderthal成分,This is consistent with earlier studies that suggest additional archaic admixture events in the ancestors of present-day Papuans
参考资料
https://www.bilibili.com/video/BV1XQ4y1N7sx?t=1864
https://bodkan.net/admixr/articles/tutorial.html