基础概念
基因渐渗(introgression):,Introgression is the movement of a gene from one species into the gene pool of another by the repeated backcrossing of an interspecific hybrid with one of its parent species。关键点:属于两个物种之间的基因流动,通常是后代种与祖先种的反复回交实现祖先种的一些基因区段(表现为潜在的祖先性状)再次出现在后代种当中。
ABBA-BABA统计:也称为D统计量,是研究基因渐渗的经典方法。在一个给定的拓扑关系(((H1,H2),H3),H4)。H1,H2是姊妹类群(多为一个物种内的两个种群),H3是潜在的基因渐渗来源物种,H4是外类群。假设祖先H4的位点为A,潜在渐渗祖先种H3位点为B,则如果不存在H3的渐渗,则H1和H2的两个种群为AB或者BA的概率各为50%,而如果H3对H2存在有渐渗作用,则H2为B,H1为A的概率大于H2为A的概率。
以下即为计算D-统计量的方法,当D值为0时,表示H1,H2与H3不存在渐渗,当D大于0,则表示ABBA的情况更多,表示H2和H3之间的关系相对于H1更近,即存在于H3到H2的渐渗。D值范围-1~1。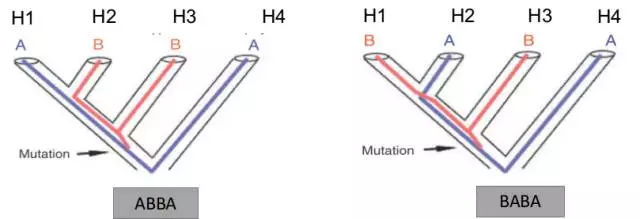
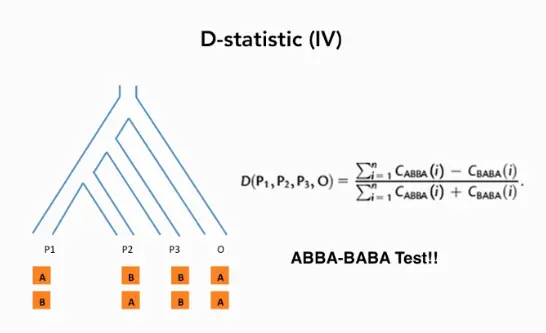
其它统计量还包括:partition D-statistic, 以及近期较为流行的fd统计值,fdm统计值等。
数据准备和解释
- 教程数据来源于3个物种multi种群和1个外类群物种的全基因组数据(whole-genome sequencing Data)
3个物种Hm, Ht, Hc。这三个物种下的多个种群之间存在着同域物种分布(sympatry),故推测存在有两个物种种群之间的杂交渐渗。 - 数据为高深度WGS数据,已经过过滤得到双位点的SNP位点。特定的,数据仅包括18号染色体的数据,已知此染色体上的翅膀花纹颜色基因。
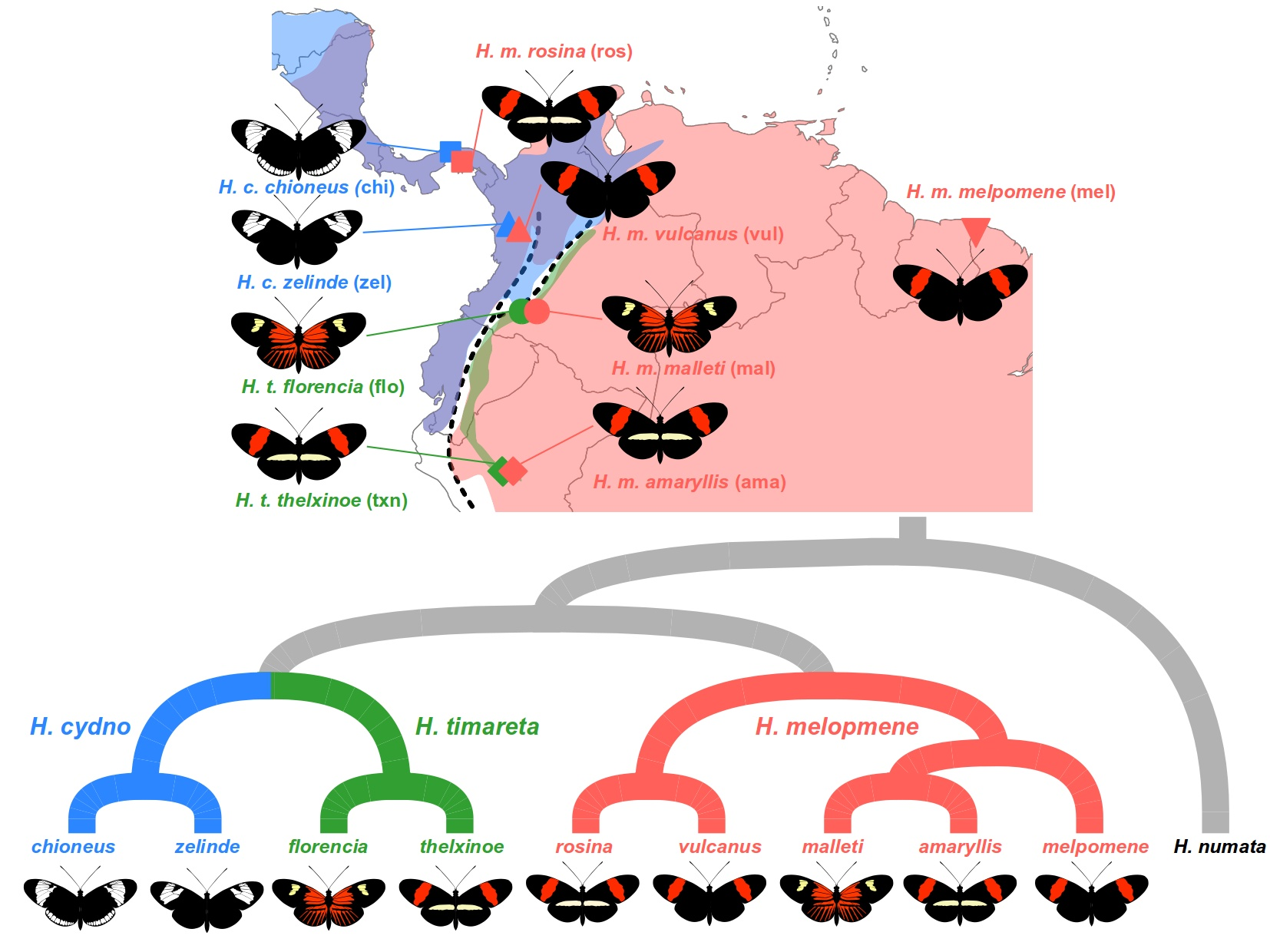
- 推测处于同一区域的两个物种之间存在有渐渗造成的基因流(sharing of genetic variation),例如Hc-chi和Hm-ros等等。已知位于18号染色体上的花纹基因optix 受到强烈选择。在一个区域的物种或通过趋同演化(independent convergent evolution)或者适应性的渐渗作用(adaptive introgression)获得此适应性的optix基因。
实操
1. 数据准备
1 | wget https://github.com/simonhmartin/tutorials/raw/master/ABBA_BABA_windows/data/hel92.DP8HET75MP9BIminVar2.chr18.geno.gz |
2. 滑动窗口检测Sliding window analysis
教程中的情况为(((P1,P2),P3),O),P1为Hm.mel(mel_mel),检测在巴拿马地区的P2(mel_ros)是否受到同域分布的P3(cyd_chi)渐渗影响;在秘鲁地区的P2(mel_ama)是否受到P3(tim_txn)的渐渗作用两种情况的外类群O均为H.num。
ABBABABAwindows.py程序基本参数
-g基因型geno文件。-f基因型文件的类型phased,pairs,haplo,diplo-P1, P2, P3, P4种群的名称--popFile:样本-种群的说明文件-wwindow大小-s步移的大小-m窗口最少位点数-o输出文件
1 | ### 巴拿马地区的渐渗检测 |
- 输出结果已包括计算的D值、fd值、fdM值
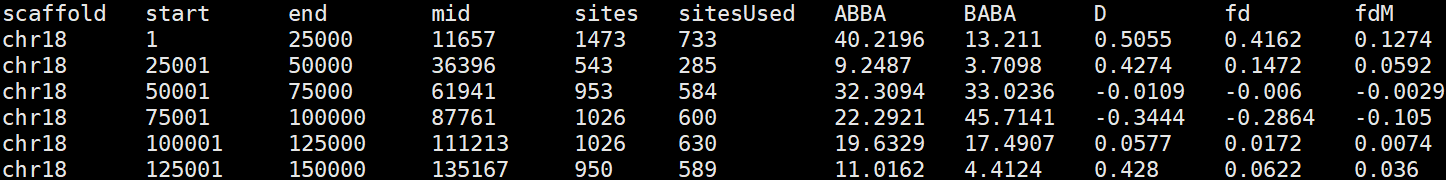
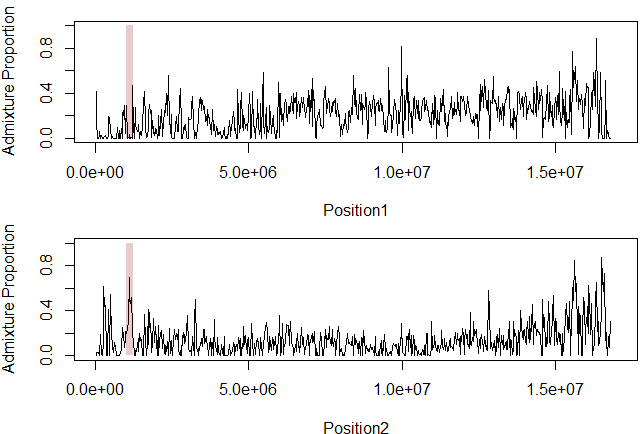
3. 简单绘图显示D值、fd值
- 读取文件
1 |
|
- 注意,如果D值为负,则fd值是无意义的。故设置所有fd值为0,当D值为负数时。
1 | for (x in 1:length(AB_tables)){ |
- 简单绘图
1 |
|
- 结果解释:总体显示,在此18号染色体上存在显著的introgression。特定的,仅观察色斑基因optix附近区域,在巴拿马地区cyd_chi对mel_ros的渐渗率很低,这也体现在二者的色斑相差很大;而在秘鲁地区的tim_txn对mel_ama渐渗率很高,这也体现在二者有相似的色斑颜色。此趋同演化现象即是渐渗作用引起的。
Refs
https://github.com/simonhmartin/tutorials/blob/master/ABBA_BABA_windows/ (多篇NG文献直接引用此教程方法)
http://www.360doc.com/content/19/0403/22/52645714_826276681.shtml